近日,我校生命科学学院副教授刘敏和讲师朱家富为论文的共同第一作者,董志诚教授为通讯作者,在植物学权威期刊Plant Cell发表论文《Comparative analysis of nascent RNA sequencing methods and their applications in studies of co-transcriptional splicing dynamics》。这是我校首次以第一单位在植物学权威期刊Plant Cell发表论文。
分子遗传与进化创新研究中心董志诚课题组长期从事植物转录的研究。在模式拟南芥中建立了GRO-seq(通过细胞核run-on在体外亲和标记新生RNA)和pNET-seq(分离RNA聚合酶II结合的RNA)的方法(Nature Plants, 2018);利用新生RNA测序方法,揭示了植物在转录水平对高温胁迫的快速响应及胁迫记忆(JIPB, 2021);在异源六倍体小麦基因组中,利用新生RNA测序技术首次检测到植物增强子的转录(Genome Biology, 2022; Star Protocols, 2022)。其中2022年在Genome Biology发表的《Enhancer transcription detected in the nascent transcriptomic landscape of bread wheat》和在Star Protocols发表的《Protocol for affordable and efficient profiling of nascent RNAs in bread wheat using GRO-seq》也均为我校首次以第一单位发表。
上述几种方法仅在少数实验室开展,大部分植物领域的实验室因为对新生RNA方法的技术特点比较陌生,难以根据实验目的和实验条件选择合适的方法,仍然依赖较为间接的mRNA-seq手段。
转录是中心法则的第一步,是传递遗传信息的关键化学反应,也是植物调整自身适应内外部环境的关键步骤,受到植物领域广泛关注。对转录产物的检测(新生RNA测序),是研究转录过程的最直接手段。然而由于相应方法缺乏,限制了植物转录的研究。寸有所长、尺有所短,哪种新生RNA方法更能够逼真地体现植物的转录反应?如何根据实验目标、可行性和成本等因素综合考虑,选择合适的方法?在这项研究中,使用了相同的植物组织、细胞核提取和尽可能相似的实验流程,以确保公平地反映每种方法的长短。
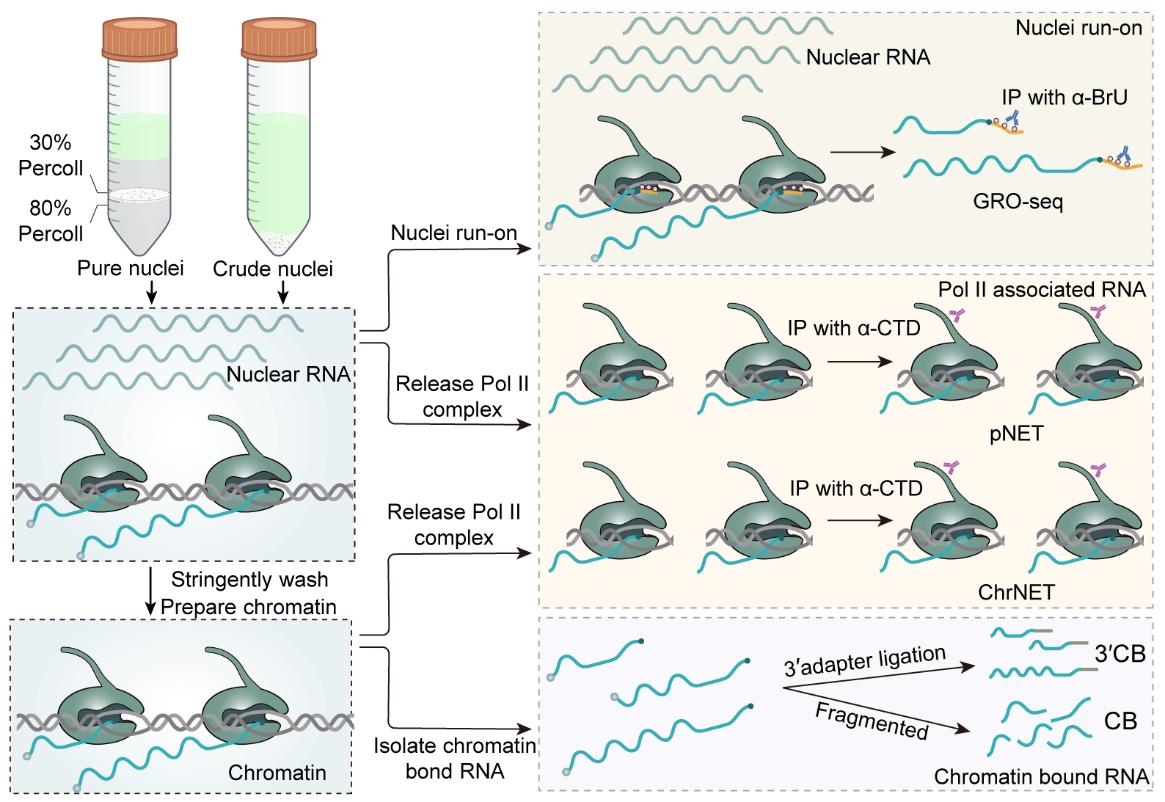
图1 不同新生RNA测序方法的比较
研究结论表明,新生RNA方法比传统mRNA-seq更灵敏地反映转录动态;pNET-seq和GRO-seq在重复性和转录细节方面表现最优;染色质RNA是很好的新生RNA代表,CB RNA-seq成本低操作简单,性价比高;3’CB RNA-seq在检测共转录剪接时最为灵敏和精确;优化后的pNET-seq方法,ChrNET在提高信噪比基础上降低了成本,优先推荐。
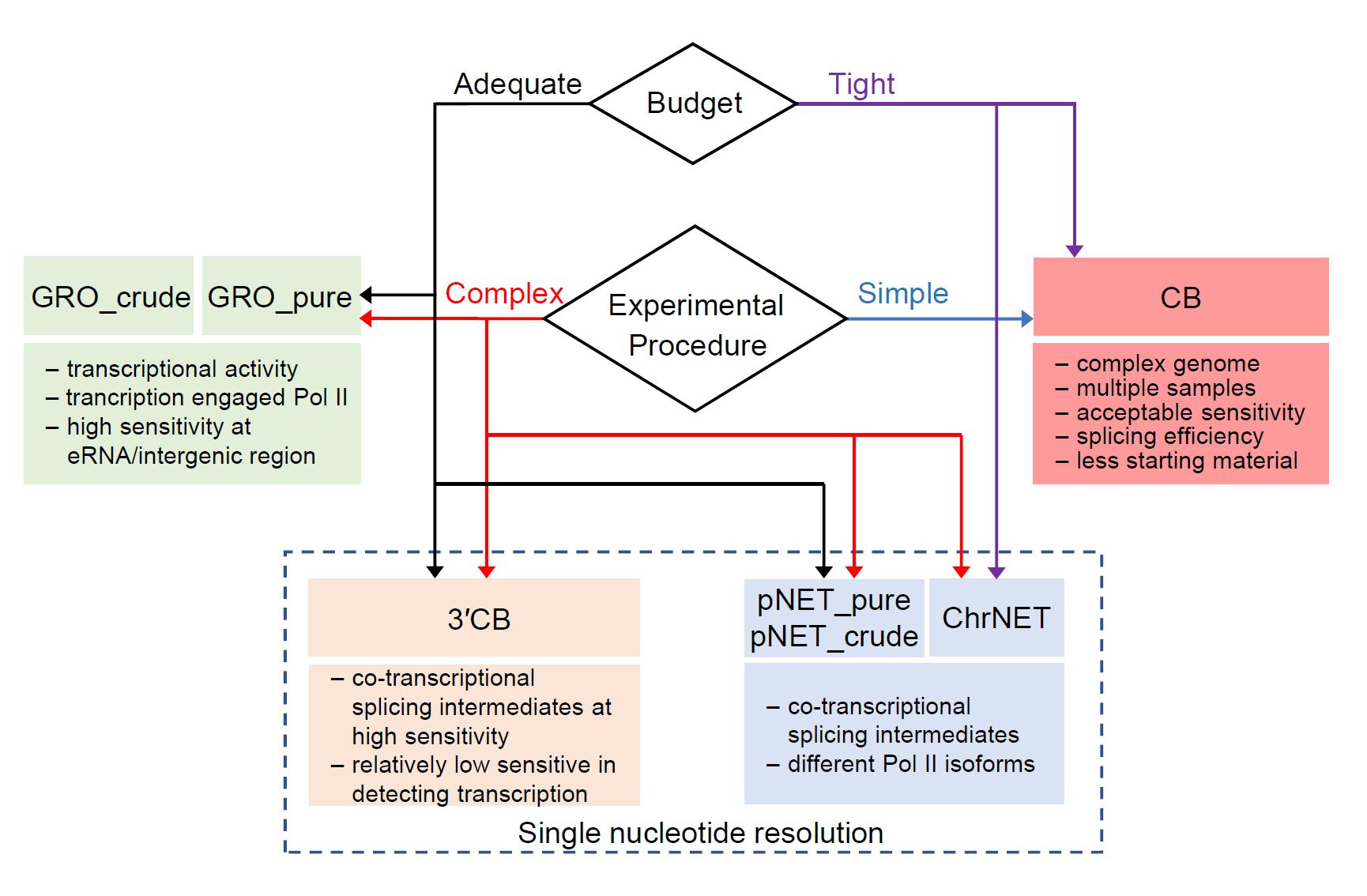
图2 新生RNA测序方法选择指引图
该研究受到了国家自然科学基金、国家重点研发、广东省植物适应性与分子设计重点实验室、广州市作物基因编辑重点实验室、广东省教育厅创新团队、广州市科技局市校联合项目的支持。
成果链接:
https://doi.org/10.1093/plcell/koad237
参考文献:
1. Min Liu#, Jiafu Zhu#, Huijuan Huang, Yan Chen, Zhicheng Dong* (2023) Comparative analysis of nascent RNA sequencing methods and their applications in studies of co-transcriptional splicing dynamics. The Plant Cell, koad237, https://doi.org/10.1093/plcell/koad237
2. Yilin Xie#, Yan Chen#, Zijuan Li#, Jiafu Zhu, Min Liu, Yijing Zhang* and Zhicheng Dong* (2022) Enhancer transcription detected in the nascent transcriptomic landscape of bread wheat. Genome Biology 23:109 10.1186/s13059-022-02675-1
3. Yan Chen#, Jiafu Zhu#, Yilin Xie, Zijuan Li, Yijing Zhang, Min Liu* and Zhicheng Dong* (2022) Protocol for affordable and efficient profiling of nascent RNAs in bread wheat using GRO-seq. Star Protocols 3(3):101657. doi: 10.1016/j.xpro.2022.101657.
4. Min Liu#, Jiafu Zhu#, Zhicheng Dong* (2021) Immediate transcriptional responses of Arabidopsis leaves to heat shock. Journal of Integrative Plant Biology 63(3): 468-483
5. Jiafu Zhu#, Min Liu#, Xiaobin Liu and Zhicheng Dong* (2018) RNA polymerase II activity revealed by GRO-seq and pNET-seq in Arabidopsis. Nature Plants 4, 1112-1123
(供稿:生命科学学院)